(Si vous trouvez trop pénible la lecture de cet article scientifique, allez directement au résumé)
La pandémie de COVID-19, causée par le SRAS-CoV-2, continue de faire rage dans de nombreux pays, mettant à rude épreuve les systèmes de santé et les économies. Les vaccins protègent contre les maladies graves et la mort et sont considérés comme essentiels pour mettre fin à la pandémie.
Les vaccins COVID-19 (et l'infection par le SRAS-CoV-2) provoquent des anticorps qui sont dirigés contre la protéine de pointe virale (S) et neutralisent le virus. Cependant, l'émergence de variantes du SRAS-CoV-2 avec des mutations de la protéine S qui confèrent une résistance à la neutralisation pourrait compromettre l'efficacité du vaccin [ 1]. De plus, des variantes virales émergentes avec une transmissibilité améliorée, probablement en raison d'interactions modifiées entre le virus et la cellule hôte, pourraient se propager rapidement à l'échelle mondiale. Par conséquent, il est important de déterminer si les variantes émergentes du SRAS-CoV-2 présentent des interactions cellulaires hôtes altérées et une résistance à la neutralisation médiée par les anticorps.
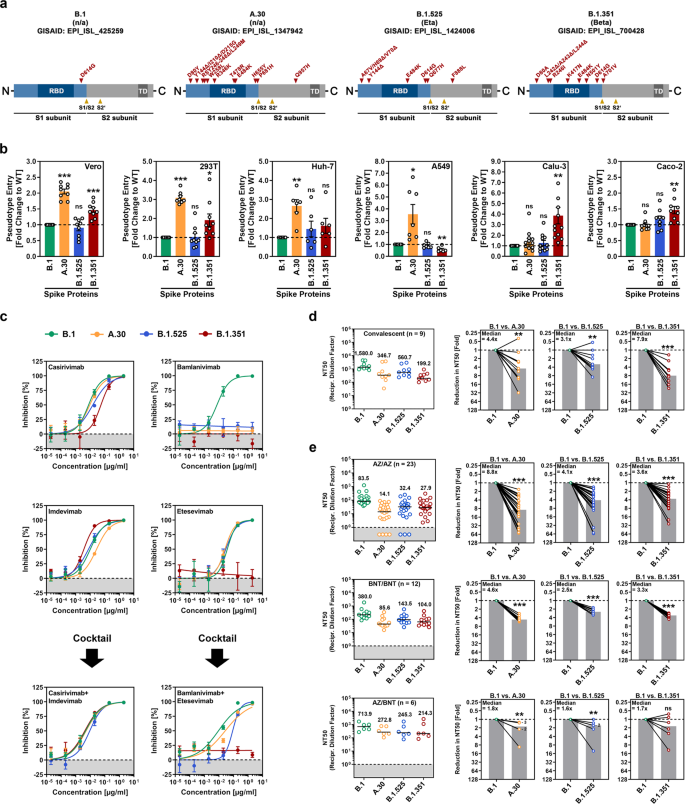
Nous avons étudié l'entrée dans la cellule hôte et la neutralisation par les anticorps du variant A.30 (également appelé A.VOI.V2), qui a été détecté chez plusieurs patients en Angola et en Suède au printemps 2021 et probablement originaire de Tanzanie [ 2 ]. A titre de comparaison, nous avons analysé les variantes Beta (B.1.351) et Eta (B.1.525). Ces deux variantes ont été détectées pour la première fois en Afrique, et la variante bêta, qui est considérée comme une variante préoccupante (COV), montre le plus haut niveau de résistance à la neutralisation parmi les COV du SARS-CoV-2 [ 3 , 4 ]. Par rapport à la protéine S du SRAS-CoV-2 B.1, qui a circulé au début de la pandémie, la protéine S du variant A.30 contient 10 substitutions d'acides aminés et cinq délétions (Fig. 1a et Informations supplémentaires, Figure. S1a ). Toutes les délétions ainsi que quatre substitutions se trouvent dans le domaine N-terminal de l'unité de surface S1, qui abrite un supersite antigénique ciblé par la plupart des anticorps neutralisants non dirigés contre le domaine de liaison au récepteur (RBD) [ 5 ]. De plus, trois mutations sont localisées à l'intérieur du RBD, qui se lie au récepteur cellulaire ACE2 et constitue la cible principale des anticorps neutralisants (Fig. 1a ). Deux de ces mutations, T478R et E484K, sont situées à proximité du site de liaison ACE2 (Informations supplémentaires, Fig. S1a), et E484K est connu pour réduire la sensibilité à la neutralisation médiée par les anticorps. Enfin, deux mutations sont localisées à proximité du site de clivage S1/S2 et une mutation se trouve dans l'unité transmembranaire S2, ce qui facilite la fusion de l'enveloppe virale avec les membranes cellulaires (Fig. 1a ).
Le SARS-CoV-2 A.30 pénètre dans certaines lignées cellulaires avec une efficacité accrue et échappe à la neutralisation médiée par les anticorps. a Aperçu schématique et organisation du domaine des protéines SARS-CoV-2 S étudiées. Abréviations : RBD, domaine de liaison au récepteur ; TD, domaine transmembranaire. bDes particules pseudotypées portant les protéines S indiquées ont été inoculées sur différentes lignées cellulaires, et l'efficacité de la transduction a été quantifiée en mesurant l'activité de la luciférase codée par le virus dans les lysats cellulaires 16 à 18h après l'inoculation. Les données moyennes (moyennes) de six à 12 réplicats biologiques (chacun réalisé avec des quadruples techniques) sont présentées pour lesquelles la transduction a été normalisée contre le SRAS-CoV-2 S B.1 (= 1). Les barres d'erreur indiquent l'erreur standard de la moyenne (SEM). La signification statistique des différences entre B.1 et A.30, B.1.525 ou B.1.351 a été analysée par le test t de Student bilatéral avec correction de Welch ( p > 0,05, non significatif [ns] ; p 0,05, * ; p 0,01, ** ; p 0,001, ***). Voir également Informations supplémentaires, Fig. S1b . c Neutralisation du SARS-CoV-2 B.1, A.30, B.1.525 et B.1.351 par des anticorps monoclonaux utilisés pour la thérapie COVID-19 ou un anticorps de contrôle non apparenté (Informations supplémentaires, Fig. S1d). Les particules pseudotypées ont été incubées pendant 30 min à 37 °C en présence de concentrations croissantes (0,0002, 0,0002, 0,002, 0,02, 0,2, 2 µg/ml) des anticorps monoclonaux indiqués ou d'un anticorps témoin non apparenté avant d'être inoculées sur des cellules Vero. L'efficacité de l'infection a été quantifiée en mesurant l'activité de la luciférase codée par le virus dans les lysats cellulaires 16 à 18 h après l'inoculation. Sont présentées les données moyennes (moyennes) d'un seul réplicat biologique (réalisé avec des quadruples techniques) pour lesquels l'infection a été normalisée par rapport à des échantillons qui ne contenaient pas d'anticorps (= 0% d'inhibition). Les données ont été confirmées dans une expérience indépendante distincte. Les barres d'erreur indiquent l'écart type. réNeutralisation du SARS-CoV-2 B.1, A.30, B.1.525 et B.1.351 par des anticorps dans le plasma convalescent. Des particules pseudotypées portant les protéines S indiquées ont été incubées pendant 30 min en présence de différentes dilutions de plasma convalescent ( n = 9). L'efficacité de l'infection a été déterminée comme décrit pour la figure 1b et utilisée pour calculer le facteur de dilution plasmatique conduisant à une réduction de 50 % de l'entrée des cellules induite par la protéine S (titre neutralisant 50, NT50). Les données d'un total de neuf échantillons de plasma de convalescent sont présentées (les lignes noires et les valeurs numériques indiquent le NT50 médian). De plus, pour chaque plasma, le facteur de réduction du NT50 entre le SARS-CoV-2 B.1 (défini comme 1) et les variantes indiquées a été calculé (les barres grises indiquent la médiane). La signification statistique des différences entre les groupes indiqués a été analysée par un test bilatéral de Mann-Whitney avec un niveau de confiance de 95 % ( p > 0,05, ns ; p 0,05, * ; p 0,01, ** ; p ≤ 0,001, ***). eL'expérience a été réalisée comme décrit dans le panneau d, mais le sérum de ChAdOx1 nCoV-19/ChAdOx1 nCoV-19 (AZ/AZ ; n = 23), BNT162b2/BNT162b2 (BNT/BNT ; n = 12) ou ChAdOx1 nCoV-19/ Des individus vaccinés par BNT162b2 (AZ/BNT ; n = 6) ont été étudiés. Les chiffres dans les graphiques à barres « B.1 vs. A.30 » et « B.1 vs. B.1.525 » indiquent le nombre de points de données qui se chevauchent (points)
Pour l'analyse de l'entrée virale dans les cellules et de son inhibition par les anticorps, nous avons utilisé des pseudotypes rhabdoviraux portant la protéine SARS-CoV-2 S, un modèle adéquat pour étudier l'entrée et la neutralisation du SARS-CoV-2 [ 6 ]. Comme cibles, nous avons utilisé les lignées cellulaires Vero et 293T (dérivées du rein), Huh-7 (foie), A549 (poumon), Calu-3 (poumon) et Caco-2 (côlon). B.1 est entré efficacement dans toutes les lignées cellulaires, et l'efficacité d'entrée de B.1.525 était comparable (Fig. 1b et Informations supplémentaires , Fig. S1b ). L'entrée de B.1.351 dans plusieurs lignées cellulaires a été légèrement mais significativement augmentée, et ce phénotype était particulièrement robuste pour les cellules pulmonaires Calu-3, conformément aux résultats publiés [ 3]. Enfin, A.30 a montré une efficacité nettement accrue concernant l'entrée dans les cellules Vero, 293 T, Huh-7 et A549 par rapport à B.1, bien que l'entrée dans les cellules Calu-3 et Caco-2 n'ait pas été augmentée (Fig. 1b ). Les tests d'anticorps monoclonaux dirigés contre la protéine S et utilisés pour la thérapie COVID-19 ont révélé que B.1.351 était résistant à la fois au bamlanivimab et à l'etesevimab, comme prévu [ 3 ] et que B.1.525 était résistant au bamlanivimab (Fig. 1c ). A.30 était également résistant au bamlanivimab mais sensible à l'inhibition par un cocktail de bamlanivimab et d'etesevimab (Fig. 1c ). De plus, B.1.351 a montré une neutralisation nettement réduite par les anticorps induits lors de l'infection, comme prévu ; [ 3] l'évasion de neutralisation par A.30 et B.1.525 était légèrement (A.30) à modérément (B.1.525) moins efficace (Fig. 1d et Informations supplémentaires, Fig. S2 ). A l'inverse, A.30 était plus résistant à la neutralisation par les anticorps induits lors de la vaccination homologue ChAdOx1 nCoV-19 (Vaxzevria) ou BNT162b2 (Comirnaty) par rapport à B.1.351, mais la sensibilité à la neutralisation de B.1.525 était approximativement dans la même plage que celle de B.1.351 (Fig. 1e et Informations supplémentaires, Fig. S2 et Tableau S1 ). Enfin, toutes les variantes testées présentaient une évasion réduite et comparable des anticorps induite par la vaccination hétérologue ChAdOx1 nCoV-19/BNT162b2, conformément aux résultats publiés pour la variante Delta (B.1.617.2) [7 ].
En résumé, A.30 présente une préférence de lignée cellulaire non observée pour les autres variantes virales et échappe efficacement à la neutralisation par les anticorps induits par la vaccination ChAdOx1 nCoV-19 ou BNT162b2. L'entrée du SRAS-CoV-2 dans les lignées cellulaires dépend de l'activation de la protéine S par les protéases cellulaires cathepsine L ou TMPRSS2 [ 8 ], et on pense que l'activation par cette dernière favorise la propagation virale dans les poumons. Par conséquent, il convient de noter qu'une entrée A.30 améliorée a été observée pour les lignées cellulaires avec la cathepsine L (Vero, 293 T, Huh-7, A549) - mais pas l'entrée dépendante de TMPRSS2 (Calu-3, Caco-2) [ 8 ]. Ainsi, on pourrait supposer que A.30 pourrait utiliser la cathepsine L avec une efficacité accrue et une résistance légère (mais non statistiquement significative) de A.30 contre l'inhibiteur de la cathepsine L MDL 28170 soutient cette possibilité (Informations supplémentaires, Fig. S1c ). Notamment, une entrée robuste dans les lignées cellulaires a été combinée à une résistance élevée aux anticorps induits lors de la vaccination ChAdOx1 nCoV-19 ou BNT162b2. La résistance à la neutralisation a dépassé celle du variant Beta (B.1.351), qui est nettement résistant à la neutralisation en culture cellulaire et, par rapport au variant Alpha (B.1.1.7), est moins bien inhibé par le vaccin ChAdOx1 nCoV-19 [ 9 ]. Néanmoins, la vaccination ChAdOx1 nCoV-19/BNT162b2 hétérologue, dont il a été précédemment démontré qu'elle augmentait les réponses en anticorps neutralisants contre les COV par rapport aux vaccinations homologues correspondantes [ 7 , 10], pourrait offrir une protection robuste contre la variante A.30. Collectivement, nos résultats suggèrent que la variante A.30 du SRAS-CoV-2 peut échapper au contrôle par les anticorps induits par le vaccin et pourrait montrer une capacité accrue à pénétrer dans les cellules d'une manière dépendante de la cathepsine L, ce qui pourrait particulièrement aider à la propagation extrapulmonaire. En conséquence, la propagation potentielle du variant A.30 justifie une surveillance étroite et la mise en place rapide de contre-mesures.
Les références
- 1.
Harvey WT, Carabelli AM, Jackson B, Gupta RK, Thomson EC, Harrison EM, et al. Variantes du SRAS-CoV-2, mutations de pointe et évasion immunitaire. Nat Rev Microbiol. 2021;19:409-24.
- 2.
COV-Lineages.org. Lignée A.30, https://cov-lineages.org/lineage.html?lineage=A.30 (2021).
- 3.
Hoffmann M, Arora P, Groß R, Seidel A, Hörnich BF, Hahn AS, et al. Les variantes B.1.351 et P.1 du SRAS-CoV-2 s'échappent des anticorps neutralisants. Cellule. 2021;184:2384-93.
- 4.
Wibmer CK, Ayres F, Hermanus T, Madzivhandila M, Kgagudi P, Oosthuysen B, et al. Le SARS-CoV-2 501Y.V2 échappe à la neutralisation par le plasma d'un donneur sud-africain COVID-19. Nat Med. 2021;27:622-5.
- 5.
McCallum M, De Marco A, Lempp FA, Tortorici MA, Pinto D, Walls AC, et al. La cartographie antigénique du domaine N-terminal révèle un site de vulnérabilité pour le SRAS-CoV-2. Cellule. 2021;184:2332–47.
- 6.
Schmidt F, Weisblum Y, Muecksch F, Hoffmann HH, Michailidis E, Lorenzi JCC, et al. Mesure de l'activité des anticorps neutralisants SARS-CoV-2 à l'aide de virus pseudotypés et chimériques. J Exp Med. 2020;217:e20201181.
- 7.
Behrens GM, Cossmann A, Stankov MV, Nehlmeier I, Kempf A, Hoffmann M, et al. Neutralisation du variant delta du SRAS-CoV-2 après vaccination hétérologue ChAdOx1-S/BNT162b2. Lancette. 2021;398:1041-2.
- 8.
Hoffmann M, Kleine-Weber H, Schroeder S, Krüger N, Herrler T, Erichsen S, et al. L'entrée des cellules SARS-CoV-2 dépend de ACE2 et TMPRSS2 et est bloquée par un inhibiteur de protéase cliniquement prouvé. Cellule. 2020;181:271-80.
- 9.
Madhi SA, Baillie V, Cutland CL, Voysey M, Koen AL, Fairlie L, et al. Efficacité du vaccin ChAdOx1 nCoV-19 Covid-19 contre la variante B.1.351. N. Engl J Med. 2021;384:1885–98.
- dix.
Barros-Martins J, Hammerschmidt SI, Cossmann A, Odak I, Stankov MV, Morillas Ramos G, et al. Réponses immunitaires contre les variants du SRAS-CoV-2 après vaccination ChAdOx1 nCoV-19/BNT162b2 hétérologue et homologue. Nat Med. 2021;27:1525-9.
Remerciements
SP reconnaît le financement du BMBF (01KI2006D, 01KI20328A, 01KI20396, 01KX2021), du ministère des Sciences et de la Culture de Basse-Saxe (14-76103-184, MWK HZI COVID-19) et de la Fondation allemande pour la recherche (DFG ; PO 716/11- 1, OREN 716/14-1). MSW a reçu un financement sans restriction de Sartorius AG, Lung Research. H.-MJ a reçu des financements du BMBF (01KI2043, NaFoUniMedCovid19-COVIM : 01KX2021), du ministère d'État bavarois des Sciences et des Arts et de la Deutsche Forschungsgemeinschaft (DFG) via les groupes de formation à la recherche RTG1660 et TRR130. GB reconnaît le financement du Centre allemand de recherche sur les infections (n° de subvention 80018019238).
Le financement
Financement Open Access activé et organisé par Projekt DEAL.
Informations sur l'auteur
Affiliations
Contributions
SP et MH ont conçu le projet. PA et MH ont conçu les expériences. SP a écrit le manuscrit, tous les auteurs ont révisé le manuscrit. PA, CR, AK, LG ont réalisé les expériences. PA, MH, SP ont analysé les données. MSW, ML, SS, HMJ, MS, GB ont fourni les réactifs essentiels.
Auteurs correspondants
Déclarations éthiques
Intérêts concurrents
Les auteurs ne déclarent aucun intérêt concurrent.
Aucun commentaire:
Enregistrer un commentaire
Remarque : Seul un membre de ce blog est autorisé à enregistrer un commentaire.