Beaucoup s'inquiètent, vaccinés ou non, de savoir comment détruire la protéine de pointe du covid 19 et celles injectées dans les vaccins. Une étude japonaise intitulée "Effet dégradant de la nattokinase sur la protéine de pointe du SRAS-CoV-2" sur 12 hommes japonais, jeunes et en bonne santé, a révélé que déjà une dose unique de 2000 FU de "nattokinase" active plusieurs voies fibrinolytiques et antithrombotiques. Pour ceux qui veulent lire cette étude scientifique :
Résumé
La maladie à coronavirus 2019 (COVID-19), causée par
le coronavirus 2 du syndrome respiratoire aigu sévère (SARS-CoV-2), est
apparue comme une pandémie et a infligé d'énormes dommages à la vie des
personnes et à l'économie de nombreux pays du monde. Cependant, les agents thérapeutiques contre le SRAS-CoV-2 restent flous. Le SRAS-CoV-2 possède une protéine de pointe (protéine S), et le clivage de la protéine S est essentiel à l'entrée virale. La nattokinase est produite par Bacillus subtilis var. nattoet est bénéfique pour la santé humaine. Dans cette étude, nous avons examiné l'effet de la nattokinase sur la protéine S du SRAS-CoV-2. Lorsque
les lysats cellulaires transfectés avec la protéine S ont été incubés
avec la nattokinase, la protéine S a été dégradée de manière dépendante
de la dose et du temps. L'analyse
par immunofluorescence a montré que la protéine S à la surface des
cellules était dégradée lorsque la nattokinase était ajoutée au milieu
de culture. Ainsi, nos
résultats suggèrent que la nattokinase présente un potentiel
d'inhibition de l'infection par le SRAS-CoV-2 via la dégradation de la
protéine S.
1. Introduction
La maladie à coronavirus 2019 (COVID-19), causée par le
coronavirus 2 du syndrome respiratoire aigu sévère (SRAS-CoV-2), s'est
propagée dans le monde entier. La pandémie de COVID-19 a touché plus de 437 millions de personnes et causé plus de 6,3 millions de décès ( https://covid19.who.int/, consulté le 4 juillet 2022). L'entrée
du SRAS-CoV-2 dans les cellules hôtes est médiée par la protéine de
pointe transmembranaire (protéine S), qui forme des homotrimères qui
s'étendent à partir de l'enveloppe virale. La
protéine S est transformée et activée par des protéases cellulaires,
notamment la protéine sérine transmembranaire 2 (TMPRSS2), la cathepsine
et la furine. Il comprend deux sous-unités fonctionnelles, S1 et S2 ; la
sous-unité S1 du SRAS-CoV-2 initie la liaison virus-récepteur en
interagissant avec l'enzyme de conversion de l'angiotensine 2 (ACE2) du
récepteur de la cellule hôte humaine, et la sous-unité S2 participe à la
fusion virale avec la cellule cible, permettant l'entrée virale [ 1 ] . Le domaine de liaison au récepteur (RBD) dans la sous-unité S1 est responsable de la liaison à l'ACE2. Le clivage de la protéine S se produit à la frontière entre les sous-unités S1 et S2.
Actuellement, de nombreux pays sont impliqués dans le
développement de vaccins pour se protéger contre l'infection par le
SRAS-CoV-2, ainsi, le nombre d'infections par le SRAS-CoV-2 a diminué. Cependant,
de nombreuses variantes du SRAS-CoV-2, y compris des souches avec des
épitopes cibles vaccinaux mutés, ont été signalées [ 2 , 3 ]. La
vaccination peut ne pas protéger complètement contre l'infection par le
SRAS-CoV-2 car le nombre de patients atteints de COVID-19 augmente
après la vaccination. Par conséquent, il est important de développer de nouveaux traitements pour les infections par le SARS-CoV-2.
Le natto est un aliment japonais traditionnel populaire à base de soja fermenté par Bacillus subtilis var. natto . La nattokinase se trouve dans le natto [ 4 ] et est l'une des enzymes extracellulaires les plus importantes produites par B. subtilis var. natto [ 5 ]. La nattokinase est constituée de 275 acides aminés et pèse environ 28 kDa [ 6 , 7 ]. La nattokinase inactive l'inhibiteur de l'activateur du plasminogène-1 et augmente la fibrinolyse [ 8 ]. Il diminue également les taux plasmatiques de fibrinogène, de facteur VII, de cytokines et de facteur VIII [ 9]. La nattokinase a le pouvoir de dissolution des caillots le plus élevé parmi les anticoagulants naturellement connus [ 10 ]. Un essai clinique a démontré que la consommation orale de nattokinase n'était associée à aucun effet indésirable [ 11 ]. Ainsi,
la nattokinase est maintenant considérée comme une enzyme efficace,
sûre et économique qui a attiré l'attention dans les études sur les
médicaments thrombolytiques [ 12 , 13 ]. De plus, la nattokinase est utilisée dans le traitement de certaines tumeurs [ 14 , 15 ].
Une étude récente a révélé que l'extrait de natto
inhibe l'herpèsvirus bovin 1 (BHV-1) et l'infection par le SRAS-CoV-2 [ 16 ]. Ces résultats indiquent que la protéase d'extrait de natto pourrait être efficace contre l'infection par le SRAS-CoV-2. Dans
cette étude, nous avons cherché à déterminer si l'inhibition de
l'infection par le SRAS-CoV-2 par l'extrait de natto est causée par la
nattokinase dérivée de B. subtilis var. natto .
2. Résultats et discussion
2.1. Effets dégradants de la nattokinase sur la protéine de pointe du SRAS-CoV-2 in vitro
Nous avons d'abord étudié si la nattokinase dans
l'extrait de natto pouvait dégrader la protéine S du SRAS-CoV-2. La
protéine S du SRAS-CoV-2 joue un rôle important dans le récepteur ACE2
de la cellule hôte au cours des premiers stades de l'infection [ 17 ]. Après
avoir mélangé le lysat cellulaire d'expression de la protéine S avec
une série de dilutions de 4 fois de nattokinase (32 µg/mL, 8 µg/mL, 2
µg/mL, 500 ng/mL, 125 ng/mL, 31,25 ng/mL et 7,8125 ng/mL), Western blot a
été réalisé. La longueur
totale de la protéine S (sous-unités S1 et S2) et de la sous-unité S2
est apparue sous forme de bandes lorsque le lysat de cellule
d'expression de la protéine S a été incubé avec du D-PBS à des
concentrations de nattokinase de 500 ng/mL, 125 ng/mL, 31,25 ng/mL et
7,8125 ng/mL ( Figure 1 UN). Ensuite, nous avons examiné si la nattokinase dégrade la protéine S de manière dépendante du temps. Le lysat a ensuite été incubé avec 1 µg/mL de nattokinase pendant 10 à 180 min. La
protéine S du SRAS-CoV-2 a été dégradée par la nattokinase après 60 à
180 min d'incubation, mais pas après 10 et 30 min d'incubation ( Figure 1 B). Ainsi, la nattokinase a dégradé la protéine S de manière dépendante de la dose et du temps.
Pour confirmer si l'effet dégradant de la nattokinase
est dû à l'activité enzymatique, la nattokinase a été traitée par
chauffage ou avec un cocktail d'inhibiteurs de protéase. Lorsque la nattokinase a été chauffée à 100 ° C pendant 5 min, l'effet dégradant de la nattokinase a été perdu ( figure 1C , voie 6). De
plus, la perte des bandes de protéine S par la nattokinase a été
bloquée lorsque des inhibiteurs de protéase ont été ajoutés ( figure 1C , voies 4 et 5). Comparé
au cocktail inhibiteur de protéines I, au cocktail de protéines III,
composé d'AEBSF HCl (chlorhydrate de fluorure de 4-(2-aminoéthyl)
benzènesulfonyle), d'aprotinine, qui est un inhibiteur irréversible de
sérine protéase, et de leupeptine, qui est une cystéine-protéase,
clairement bloque l'activité de la nattokinase. La nattokinase a les mêmes acides aminés conservés, Ser-His-Asp (Asp32 , His 64 et Ser 221 ), qui sont des membres de la famille subtilisine des sérine protéases [ 6 , 18 ]. La structure cristalline de la nattokinase est presque identique à celle de la subtilisine E de B. subtilis DB104 [ 19 ]. Ce résultat est cohérent avec celui d'un rapport précédent selon lequel la nattokinase est une sérine protéase. Nous avons également évalué les effets dégradants de la nattokinase à l'aide de lysats cellulaires exprimant le RBD et l'ACE2. Lorsque 7,5 µg/mL de nattokinase et de lysat cellulaire ont été incubés, les bandes de RBD et ACE2 ont été perdues ( Figure 1 D).
2.2. Effets dégradants de la nattokinase sur la protéine de pointe du SRAS-CoV-2 à la surface des cellules transfectées
Ensuite, nous avons examiné si la nattokinase dégrade la protéine S sur la surface cellulaire transfectée. La protéine S a été transfectée avec les cellules HEK293. Les cellules transfectées ont été incubées avec de la nattokinase pendant 9 h. La
protéine S sur la surface cellulaire a été détectée à l'aide d'un
anticorps anti-protéine S sans perméabilisation cellulaire ( Figure 2 A). La protéine S a été détectée dans les cellules transfectées. Lorsque les cellules transfectées ont été traitées avec de la nattokinase, la protéine S à la surface des cellules a diminué. Lorsque
les cellules ont été traitées avec 25 µg/mL et 2,5 µg/mL de
nattokinase, le rapport entre la zone positive pour la protéine S et la
zone positive pour le noyau a diminué d'environ 0,3 et 0,7,
respectivement ( figure 2 B). L'effet dégradant de la nattokinase a été observé en l'absence de cytotoxicité (Figure 2C). L'analyse
Western blot a montré que la quantité de protéine S totale ne changeait
pas entre les traitements de nattokinase et de contrôle ( Figure supplémentaire; Figure S1 ). Ces résultats indiquent que la nattokinase dégraderait la protéine S du SRAS-CoV-2 dans la plage de concentration non toxique.
Dans cette étude, nous avons montré que l'activité
protéasique de la nattokinase contribue à la dégradation de la protéine
S. La nattokinase a un effet dégradant non seulement sur les protéines S mais aussi sur l'ACE2 dans les cellules hôtes. La
spécificité de la protéase de la nattokinase serait faible, car GAPDH,
une protéine domestique, a également été dégradée simultanément lors de
l'évaluation in vitro de la nattokinase mélangée à un lysat cellulaire (
Figure supplémentaire; Figure S2 ). D'autre
part, lorsqu'il est ajouté aux cellules, il ne montre aucun effet sur
la viabilité cellulaire et devrait agir comme agent protecteur à la
surface des cellules. Une
analyse plus approfondie des produits de dégradation de la nattokinase à
l'aide de la spectrométrie de masse est nécessaire pour comprendre les
effets de la protéolyse.
La nattokinase possède la puissante activité de
dégradation de la protéine S du SRAS-CoV-2 et il a également été
démontré qu'elle exerce des effets anti-athéroscléreux, hypolipémiants,
antihypertenseurs, antithrombotiques, fibrinolytiques, neuroprotecteurs,
antiplaquettaires et anticoagulants [ 20 ]. Les
patients souffrant d'hypertension et de comorbidités cardiovasculaires
peuvent facilement tomber très malades à cause du COVID-19 [ 21 ]. En
raison de l'émergence de nombreuses variantes du SRAS-CoV-2, y compris
des souches avec des épitopes cibles vaccinaux mutés, la vaccination
seule peut ne pas protéger complètement contre l'infection par le
SRAS-CoV-2. Les extraits
de nattokinase et de natto ont le potentiel d'être développés en tant
que nouvelle génération de médicaments pour la prévention et le
traitement du COVID-19.
3. Matériels et méthodes
3.1. Matériaux
La nattokinase a été obtenue auprès de Contek Life Science Co., Ltd. (Taipei City, Taiwan). L'activité nattokinase était de 60 000 FU/g (FU, unité de fibrinolyse). Les
ensembles de cocktails d'inhibiteurs de protéase I et III ont été
achetés auprès de FUJIFILM Wako Pure Chemical Corporation (Osaka,
Japon). Le plasmide
d'expression (pcDNA3.1-SARS2-Spike C9 avec étiquette au C-terminal,
pcDNA3.1-hACE2 et pcDNA3-SARS-CoV-2-S-RBD-sfGFP) a été acheté chez
Addgene (Watertown, MA, ETATS-UNIS). Les cellules HEK293 (JCRB9068) ont été obtenues auprès de la JCRB Cell Bank (Osaka, Japon).
3.2. Culture cellulaire et transfert Western
Les cellules HEK293 ont été cultivées à une densité de 3,5 × 10 5
cellules/mL dans du DMEM additionné de 10 % de FBS, de L-glutamine, de
pénicilline 100 U/mL et de streptomycine 100 μg/mL pendant la nuit. Les
cellules ont été transfectées avec chaque plasmide
(pcDNA3.1-SARS2-Spike, pcDNA3-SARS-CoV-2-S-RBD-sfGFP ou pcDNA3.1-hACE2)
et incubées pendant 22 h. Après
incubation, les cellules cultivées ont été grattées et lavées avec une
solution saline tamponnée au phosphate de Dulbecco glacée (D-PBS). Le
comptage des cellules a été effectué et le tampon xTractor (Takara Bio
Inc., Shiga, Japon) a été ajouté au précipité cellulaire. Les lysats cellulaires ont été centrifugés à 1300× gpendant 10 min à 4 ° C et le surnageant a été transféré dans de nouveaux tubes et stocké à -80 ° C jusqu'à utilisation. La
concentration en protéines a été déterminée par dosage de protéines à
l'acide bicinchoninique (BCA) à l'aide d'un kit de dosage BCA (Takara). Dix microlitres de nattokinase et 10 µL de lysat cellulaire (1 µg/mL) ont été incubés à 37 °C pendant 1 h. Lorsque
les effets des inhibiteurs de protéase ont été utilisés, les ensembles
de cocktails d'inhibiteurs de protéase I et III ont été dilués 10 fois
avec du D-PBS, et une solution de cocktail d'inhibiteur de protéase de
10 µL a été ajoutée au mélange de nattokinase et de lysat cellulaire. Des volumes égaux du mélange réactionnel ont été chargés et un Western blot a été effectué. Les
anticorps primaires comprenaient l'anticorps monoclonal de souris
anti-rhodopsine (C9) (1D4) (Santa Cruz Biotechnology, Dallas, TX, USA),
l'anticorps monoclonal de souris anti-GAPDH (FUJIFILM Wako), anticorps monoclonal de souris anti-étiquette GFP (Proteintech, Rosemont, IL, USA) et anticorps anti-ACE2 (Proteintech). Les anticorps secondaires comprennent un anticorps anti-souris de chèvre conjugué à la HRP (Proteintech).
3.3. Test d'immunofluorescence
Les cellules HEK293 ont été cultivées à une densité de 3,5 × 10 5cellules/mL
dans une chambre à 8 puits dans du DMEM additionné de 10 % de FBS, de
L-glutamine, de pénicilline 100 U/mL et de streptomycine 100 μg/mL. Les cellules ont été transfectées avec pcDNA3.1-SARS2-Spike et incubées pendant 9 h. Après
incubation, les cellules ont été traitées avec les échantillons,
incubées pendant 13 h et fixées dans du paraformaldéhyde à 4 % pendant
30 min. Après incubation
avec l'anticorps monoclonal de pointe SARS-CoV/SARS-CoV-2 (1A9)
(GeneTex, CA, USA) pendant 1 h, il a été incubé avec un anticorps
anti-souris de chèvre conjugué à Cy3 pendant 1 h. Les
lames ont été colorées avec DAPI Fluoromount-G et observées à l'aide
d'un microscope à fluorescence (BZ-X710, Keyence, Osaka, Japon). Les
zones positives pour la protéine S et positives pour le noyau ont été
calculées à l'aide du logiciel d'analyse joint BZ-X710 (analyseur BZ-X).
La viabilité cellulaire a
été évaluée à l'aide du test de bromure de
3-(4,5-diméthylthiazol-2-yl)-2,5-diphényltétrazolium (MTT). Les cellules ont été cultivées dans des plaques de culture à 24 puits. Après incubation à 37 °C pendant 24 h, des échantillons ont été ajoutés à chaque puits et incubés pendant 13 h supplémentaires. Les cellules ont été mises en suspension dans 500 μL de DMEM contenant 500 μg/mL de MTT. Après
incubation pendant 3 h à 37 ° C, 500 μL d'isopropanol contenant 4 mM de
HCl ont été ajoutés pour dissoudre le formazan MTT. L'absorbance a été mesurée à 570 nm à l'aide d'un lecteur de microplaques.
4. Conclusions
Dans cette étude, nous avons démontré que la
nattokinase, une sérine protéase, dégrade la protéine S du SRAS-CoV-2. Pour
déterminer si la nattokinase contenue dans l'extrait de natto pouvait
inhiber l'infection par le SRAS-CoV-2, nous avons analysé la dégradation
de la protéine S en mélangeant le lysat de cellules d'expression de la
protéine S et la nattokinase d'une manière dépendante de la dose et du
temps. Le RBD de la protéine S se lie à la partie distale de la membrane de la protéine ACE2. Il
a été rapporté que l'extrait de natto inhibe l'infection par le
SRAS-CoV-2 dans les cellules Vero E6 via la dégradation de la RBD [ 16]. Nous
avons démontré que la dégradation de la protéine S par la nattokinase
était bloquée par des traitements thermiques ou inhibiteurs de
protéines. Nos données suggèrent que l'activité protéasique de la nattokinase joue un rôle crucial dans la dégradation de la protéine S. Pris
ensemble, ces découvertes supportent la notion que l'inhibition de
l'infection SARS-CoV-2 par l'extrait de natto était due à la dégradation
de la protéine S par la nattokinase. Ainsi,
nos données ont indiqué que les extraits de nattokinase et de natto ont
des effets potentiels sur l'inhibition de l'entrée de la cellule hôte
du SRAS-CoV-2 via la dégradation de la protéine S.
Matériel supplémentaire
Les informations complémentaires suivantes peuvent être téléchargées à l'adresse : https://www.mdpi.com/article/10.3390/molecules27175405/s1
, Figure S1 : l'effet de l'ajout de nattokinase au milieu de culture
cellulaire exprimant la protéine S a été évalué par transfert Western ; Figure S2 : Effets dégradants de GAPDH in vitro.
Les contributions de l'auteur
Conceptualisation et supervision, TT, JY, KH, SC, AI, TY, RS, YI et MK ; Méthodologie, tous auteurs; Western blot et analyses immunofluorescentes, TT, YK et MK ; rédaction—préparation du brouillon original, TT et MK ; rédaction—révision et édition, tous les auteurs. Tous les auteurs ont lu et accepté la version publiée du manuscrit.
Financement
Cette recherche n'a reçu aucun financement externe.
Déclaration du comité d'examen institutionnel
N'est pas applicable.
Déclaration de consentement éclairé
N'est pas applicable.
Déclaration de disponibilité des données
Les données utilisées pour étayer ces conclusions ont été incluses dans cet article. Des informations supplémentaires sont disponibles sur demande auprès des auteurs correspondants.
Les conflits d'intérêts
Les auteurs ne déclarent aucun conflit d'intérêt.
Références
- Alsobaie, S. Comprendre la biologie moléculaire du SRAS-CoV-2 et de la pandémie de COVID-19 : un examen. Infecter. Résistance aux médicaments. 2021 , 14 , 2259–2268. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Bayati, A.; Kumar, R.; François, V.; McPherson, PS Le SRAS-CoV-2 infecte les cellules après l'entrée virale par endocytose médiée par la clathrine. J. Biol. Chim. 2021 , 296 , 100306. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Bestle, D.; Heindl, M. ; Limburg, H.; Van Lam van, T.; Pilgram, O. ; Moulton, H.; Stein, DA; Hardes, K.; Eickmann, M.; Dolnik, O.; et coll. Le TMPRSS2 et la furine sont tous deux essentiels à l'activation protéolytique du SRAS-CoV-2 dans les cellules des voies respiratoires humaines. Vie Sci. Alliance 2020 , 3 , e202000786. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Sumi, H.; Hamada, H.; Tsushima, H. ; Mihara, H.; Muraki, H. Une nouvelle enzyme fibrinolytique (nattokinase) dans le fromage végétal Natto ; un aliment à base de soja typique et populaire dans l'alimentation japonaise. Expérientia 1987 , 43 , 1110–1111. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Dabbagh, F.; Negahdaripour, M.; Berenjian, A.; Behfar, A.; Mohammadi, F.; Zamani, M.; Irajie, C.; Ghasemi, Y. Nattokinase : Production et application. Appl. Microbiol. Biotechnol. 2014 , 98 , 9199–9206. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Nakamura, T.; Yamagata, Y.; Ichishima, E. Séquence nucléotidique du gène NAT de la subtilisine, aprN, de Bacillus subtilis (natto). Biosci. Biotechnologies. Bioch. 1992 , 56 , 1869–1871. [ Google Scholar ] [ CrossRef ] [ PubMed ]
- Urano, T.; Ihara, H.; Umemura, K.; Suzuki, Y.; Oike, M.; Akita, S.; Tsukamoto, Y.; Suzuki, I.; Takada, A. L'enzyme profibrinolytique subtilisine NAT purifiée à partir de Bacillus subtilis Clive et inactive l'inhibiteur de l'activateur du plasminogène de type 1. J. Biol. Chim. 2001 , 276 , 24690–24696. [ Google Scholar ] [ CrossRef ]
- Suzuki, Y.; Kondo, K.; Matsumoto, Y.; Zhao, BQ ; Otsuguro, K.; Maeda, T.; Tsukamoto, Y.; Urano, T.; Umemura, K. La supplémentation alimentaire en soja fermenté, natto, supprime l'épaississement de l'intima et module la lyse des thrombus muraux après une lésion endothéliale dans l'artère fémorale du rat. Vie Sci. 2003 , 73 , 1289–1298. [ Google Scholar ] [ CrossRef ]
- Hsia, CH; Shen, MC; Lin, JS; Wen, YK ; Hwang, KL; Cham, TM; Yang, NC La nattokinase diminue les taux plasmatiques de fibrinogène, de facteur VII et de facteur VIII chez les sujets humains. Nutr. Rés. 2009 , 29 , 190–196. [ Google Scholar ] [ CrossRef ]
- Weng, Y.; Yao, J.; Sparks, S.; Wang, KY Nattokinase : un agent antithrombotique oral pour la prévention des maladies cardiovasculaires. Int. J. Mol. Sci. 2017 , 18 , 523. [ Google Scholar ] [ CrossRef ]
- Kim, JY; gomme, SN; Paik, JK; Lim, HH ; Kim, KC ; Ogasawara, K.; Inoue, K.; Park, S.; Jang, Y.; Lee, JH Effets de la nattokinase sur la tension artérielle : un essai randomisé et contrôlé. Hypertens. Rés. 2008 , 31 , 1583–1588. [ Google Scholar ] [ CrossRef ]
- Wu, C.; Gao, C.; Lu, S.; Xu, X.; Wen, N.; Zhang, S.; Liu, M. Construction de nanocomposites de dendrimère de polylysine portant la nattokinase et leur application à la thrombolyse. J. Biomed. Mater. Rés. A 2018 , 106 , 440–449. [ Google Scholar ] [ CrossRef ]
- Lampe, BJ; Anglais, JC Évaluation toxicologique de la nattokinase dérivée de Bacillus subtilis var natto . Chimie alimentaire. Toxicol. 2016 , 88 , 87–99. [ Google Scholar ] [ CrossRef ]
- Kou, Y. ; Feng, R.; Chen, J.; Duan, L.; Wang, S.; Hu, Y.; Zhang, N.; Wang, T.; Deng, Y.; Song, Y. Développement d'un complexe nattokinase-acide polysialique pour le traitement avancé des tumeurs. EUR. J.Pharm. Sci. 2020 , 145 , 105241. [ Google Scholar ] [ CrossRef ]
- Yan, Y.; Wang, Y.; Qian, J.; Wu, S.; Ji, Y.; Liu, Y.; Zeng, J.; Gong, A. L'extrait brut de nattokinase inhibe la croissance du carcinome hépatocellulaire chez la souris. J. Microbiol. Biotechnol. 2019 , 29 , 1281–1287. [ Google Scholar ] [ CrossRef ]
- Oba, M.; Rongduo, W.; Saito, A.; Okabayashi, T.; Yokota, T.; Yasuoka, J.; Sato, Y.; Nishifuji, K.; Wake, H.; Nibu, Y.; et coll. L'extrait de natto, un aliment de soja fermenté japonais, inhibe directement les infections virales, y compris le SRAS-CoV-2 in vitro. Biochimie. Biophys. Rés. Commun. 2021 , 570 , 21–25. [ Google Scholar ] [ CrossRef ]
- Smaoui, M. ; Yahyaoui, H. Démêler le paysage de stabilité des mutations dans le domaine de liaison au récepteur SARS-CoV-2. Sci. Rép. 2021 , 11 , 9166. [ Google Scholar ] [ CrossRef ]
- Carter, P.; Wells, JA Dissection de la triade catalytique d'une sérine protéase. Nature 1988 , 332 , 564–568. [ Google Scholar ] [ CrossRef ]
- Yanagisawa, Y.; Chatake, T. ; Naito, S.; Ohsugi, T.; Yatagai, C.; Sumi, H.; Kawaguchi, A.; Chiba-Kamosida, K.; Ogawa, M.; Adachi, T.; et coll. Détermination de la structure aux rayons X et deutération de la nattokinase. J. Radiat synchrotron. 2013 , 20 Pt 6 , 875–879. [ Google Scholar ] [ CrossRef ]
- Chen, H.; McGowan, EM; Ren, N.; Lal, S.; Nassif, N.; Shad-Kaneez, F.; Qu, X.; Lin, Y. Nattokinase : Une alternative prometteuse dans la prévention et le traitement des maladies cardiovasculaires. Biomarque. Insights 2018 , 13 , 1177271918785130. [ Google Scholar ] [ CrossRef ]
- Cancarevic, I.; Malik, BH Infection par le SRAS-CoV-2 (COVID 19) chez les patients hypertendus et chez les patients atteints de maladie cardiaque. Curéus 2020 , 12 , e8557. [ Google Scholar ] [ CrossRef ]
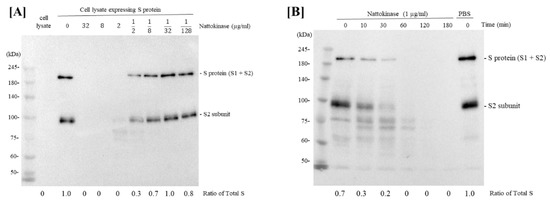
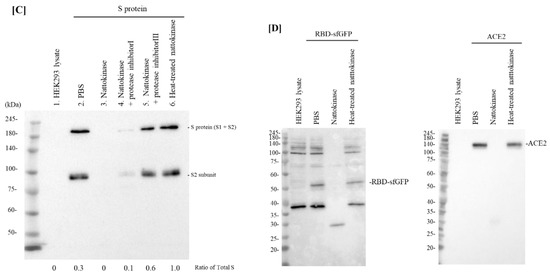
Figure 1. ( A ) Effets dégradants de la nattokinase de manière dose-dépendante. La
nattokinase diluée en série (32 µg/mL, 8 µg/mL, 2 µg/mL, 500 ng/mL, 125
ng/mL, 31,25 ng/mL et 7,8125 ng/mL) a été mélangée avec un lysat
cellulaire d'expression de la protéine S et incubée . La
longueur totale de la protéine S (sous-unités S1 et S2) et de la
sous-unité S2 a été détectée comme bandes supérieure et inférieure,
respectivement. Le rapport de S total a été indiqué comme la quantité relative de protéine S (protéine S + protéine S2). ( B ) Effets dégradants de la nattokinase de manière dépendante du temps. Le
lysat de cellules d'expression de la protéine S a été incubé avec 1
µg/mL de nattokinase pendant 0, 10, 30, 60, 120 et 180 min. ( C) Effets du traitement thermique ou des inhibiteurs de protéase. Piste 1 : lysat HEK293 ; piste 2 : lysat HEK293 (protéine S) ; piste 3 : HEK293 (protéine S) + nattokinase (5 µg/mL) ; piste 4 : HEK293 (protéine S) + nattokinase (5 µg/mL) + inhibiteur de protéase I ; piste 5 : HEK293 (protéine S) + nattokinase (5 µg/mL) + inhibiteur de protéase III ; ligne 6 : HEK293 (protéine S) + nattokinase traitée thermiquement (5 µg/mL). ( D ) Effet dégradant sur le RBD de la protéine S et de l'ACE2. La RBD de la protéine S et les plasmides codant pour l'ACE2 ont été transfectés avec des cellules HEK293, respectivement. Les
lysats cellulaires ont été incubés avec de la nattokinase (7,5 µg/mL)
et de la nattokinase traitée thermiquement (7,5 µg/mL) et un transfert
Western a été effectué.
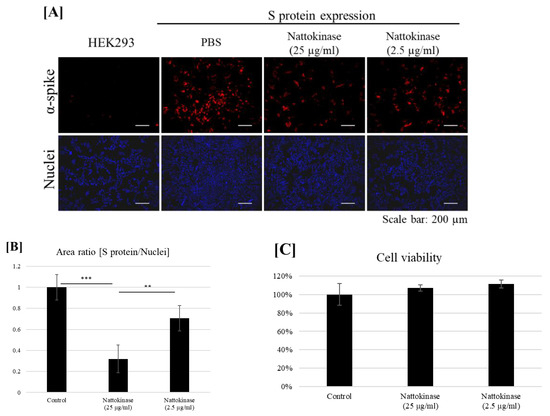
Figure 2. ( A ) Effet dégradant de la nattokinase sur la protéine S à la surface cellulaire. Spike-pcDNA3.1 a été transfecté avec des cellules HEK293 et incubé pendant 9 h. Après incubation, de la nattokinase (25 et 2,5 µg/mL) a été ajoutée au milieu de culture et encore incubée pendant 13 h. Les cellules ont été fixées et une analyse par immunofluorescence a été effectuée. La
protéine S à la surface de la cellule a été colorée avec un anticorps
anti-protéine de pointe (rouge) et le noyau a été coloré avec du DAPI
(bleu). ( B ) Rapport de la surface de la protéine S à la surface du noyau positif. Trois images par échantillon ont été capturées et les zones positives de protéine S/noyau ont été calculées. Les données sont présentées sous forme de moyenne + SD, et p-la
valeur a été déterminée par analyse de variance unidirectionnelle
(ANOVA) avec le test post-hoc de Tukey en utilisant le logiciel R
(R-3.3.3 pour les fenêtres) (** p < 0,01 ; *** p < 0,001). ( C ) La viabilité cellulaire a été évaluée par le test MTT. La nattokinase indiquée a été ajoutée au milieu de culture et incubée pendant 13 h ; Le test MTT a été
réalisé.
Source
réalisé.
Source

Aucun commentaire:
Enregistrer un commentaire
Remarque : Seul un membre de ce blog est autorisé à enregistrer un commentaire.